这个起源于群里讨论, 群里讨论到arXiv的一篇文章, 这篇文章研究称, 40%的中国人的Y染色体来自三个新石器时代的超级祖先, 在新浪博客上有比较详细的介绍.
后来群里又讨论到线粒体夏娃的事情, 来源于知乎的一个回答. 这个回答里给出了一些计算, 说明了为什么在一个有不同种群基因的种群中, 经过多代遗传, 最后都会追溯到同一个祖先.
但这里的回答给的说明不太好理解, 和群友讨论后, 我重新用python做了一个简单的计算.
假设最初种群有100名女性, 那么为了维持种群女性数量, 那么平均每名女性都要生两个孩子, 因为大约有50%的概率会生男孩.
注意!, 以下所有讨论的基因都是线粒体基因!
重复以下过程, 重复的每一次, 就是产生新的一代人:
- 产生一个由0,1组成的序列
S, 每个数字产生的概率都是50%. 这个序列代表了繁衍的下一代.
- 同时遍历这个序列
S和上一代基因G1, 如果这个序列中第i个位置的值是1, 那么保留G1[i]到G2中. 由于S的长度是大于G1的, 因此遍历的时候用取余, 即G1[i % len(G1)], S超过的部分再从G1的开头重新遍历. 这个在数学上比较优雅, 也很符合实际, 放在真实场景, 就是按年龄最大的女性生下了第一个孩子后, 到她再生第二个的时候, 差不多其他所有的女性都生过了一胎了~
- 统计上一轮产生的
G2, 看看还剩下多少独特的基因.
给出一次作图的结果, 实际上由于随机数每次产生都是不确定的, 因此每次做的图都会有些许差异:
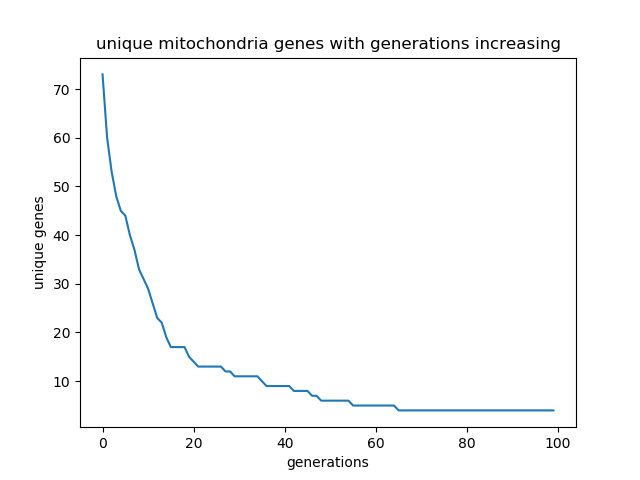
- 在种群早期, 基因的多样性锐减. 因为尽管每个母亲都会生下1个以上的孩子, 而且有50%的概率剩下女孩来传承基因, 但即使这样也只有75%的概率传承基因. 而此时基因多样性非常高, 但也意味着每个个体基因都不相同, 因此一旦没有得到传承, 这个基因会马上失效.
- 到了后期, 基因的多样性开始变得平缓. 因为种群里已经很多女性有了相同的基因了.
- 大概在100代左右的时候, 基本上剩下的基因就不到10个了. 而且其中会有一个优势基因存在, 它的数量要大于其它基因的数量, 在未来的繁衍中, 也更可能持续存在下去.
- 200代, 即2N代后, 平均会剩下2个左右的基因, 文章里说会剩下一个, 那可能是模拟的差异.
这样计算的假设是种群大小差不多不变, 这个假设并不完全合理. 因为种群数量往往是动态的, 从大的范围来讲, 这个数量是增加的, 而种群数量的增加也是有利于基因的传递的, 因为相当于每个女性可能生更多的孩子, 也就利于基因的传递.
特别的, 越在早期的时候, 即基因多样性越大的时候, 生更多的孩子会对基因多样性的传递产生更大的意义.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
|
import numpy as np
import matplotlib.pyplot as plt
import random
male = 1
female = 0
# generate children sequence
def generChildren(num):
ls = list()
for i in range(num):
k = random.randint(0, 1)
ls.append(k)
return ls
# from gen1 as mothers, generate childrenSeq children, return new genes seq
def breding(gen1, childrenSeq):
gen2 = list()
cl = len(childrenSeq)
gensLeft = len(gen1)
for i in range(cl):
if childrenSeq[i] == female:
gen2.append(gen1[i % gensLeft])
return gen2
# get statistics for genes.
def stat(gen):
d = dict()
for i in gen:
if i in d.keys():
d[i] += 1
else:
d[i] = 1
print("now have ", str(len(d)), " gens left")
return d
# NUM is approximately twice the HERD size, since there are 50% change to get a boy
NUM = 200
# HERD is initial mothers num
HERD = 100
# initial genes
ini = list()
for i in range(HERD):
# use 1,2,3,4.... as representatives for genes
ini.append(str(i))
unis = list()
for i in range(500):
c = generChildren(NUM)
newG = breding(ini, c)
ini = newG
s = stat(newG)
unis.append(len(s))
x = range(len(unis))
plt.plot(x, unis)
plt.title("unique mitochondria genes with generations increasing")
plt.xlabel("generations")
plt.ylabel("unique genes")
plt.show()
# key are genes, values is how many female with genes left
for key, values in s.items():
print(key, values)
|
